I prosessen med å utføre genetiske studier, møter vi ofte utilstrekkelige RNA-prøver, for eksempel for å studere bittesmå anatomiske orale svulster, til og med enkeltcelleprøver, og prøver av spesifikke genmutasjoner som transkriberes i svært lave nivåer i menneskeceller.Selvfølgelig, for COVID-19-testen, hvis prøvene ikke er på rett sted eller ikke nok ganger under prøvetakingen, vil prøvestørrelsen være veldig lav, og det er grunnen til at kommisjonen for helse og familieplanlegging kom ut for to dager siden og besto testen, og hvis nukleinsyreprøvetakeren ikke tok seks prøver, kan du rapportere det.
Følsomheten til reagenset er viktig fordi vi har dette eller det problemet, så hva kan vi gjøre for å forbedre følsomheten til RT-PCR?
Før vi diskuterer mulige løsninger, la oss nevne to store komplikasjoner med situasjonen vi nettopp nevnte.
Først og fremst bekymrer vi oss for RNA-tap når vi bare har noen få cellepopulasjoner i prøven vår.Dersom tradisjonelle separasjons- og rensemetoder brukes, som kolonnemetode eller nukleinsyreutfellingsmetode, er det stor mulighet for at de få prøvene går tapt.En løsning er å legge til et bærermolekyl, for eksempel tRNA, men selv da er det ingen garanti for at gjenopprettingseksperimentet vårt er OK.
Så hva er en bedre måte?Et godt alternativ for dyrkede celler eller mikroanatomiske prøver er å bruke direkte lysis.
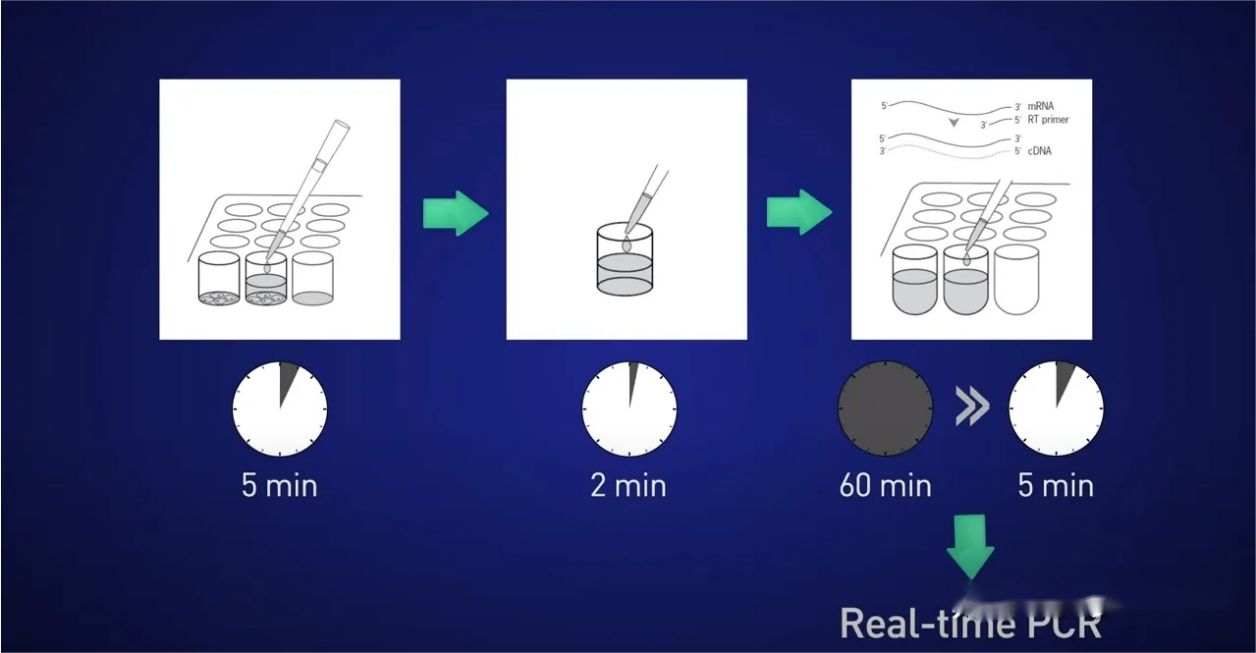
Ideen er å dele cellene i 5 minutter, frigjøre RNA i løsningen, deretter stoppe reaksjonen i 2 minutter, og deretter legge lysatet direkte til revers transkripsjonsreaksjonen slik at ingen RNA går tapt, og til slutt legge det resulterende cDNA direkte inn i sanntidsreaksjonen.
Men hva om vi, på grunn av et begrenset utgangspunkt eller en liten mengde målgenekspresjon, kan resirkulere alt RNA og fortsatt ikke gi nok maler til å få et godt sanntidssignal?
I dette tilfellet kan pre-amplifikasjonstrinnet være svært nyttig.
Følgende er et opplegg for å øke følsomheten etter revers transkripsjon.Før vi starter, må vi spørre nedstrøms hvilke mål vi er interessert i, for å designe spesifikke primere for disse målene for pre-amplifisering.
Dette kan oppnås ved å lage en blandet primer med opptil 100 par primere og en reaksjonssyklus på 10 til 14 ganger.Derfor er det nødvendig med en Master Mix spesielt designet for dette kravet for å pre-amplifisere det oppnådde cDNA.
Grunnen til å sette antall sykluser mellom 10 og 14 er at dette begrensede antallet sykluser sikrer tilfeldighet mellom de ulike målene, noe som er avgjørende for forskere som trenger kvantitativ molekylær informasjon.
Etter pre-amplifisering kan vi få en stor mengde cDNA, slik at deteksjonssensitiviteten i bakenden blir kraftig forbedret, og vi kan til og med fortynne prøven og utføre flere sanntids PCR-reaksjoner for å eliminere mulige tilfeldige feil.
Innleggstid: 11-apr-2023